Container-Software zu verwenden kann in vielen Anwendungsfällen hilfreich sein. Mit Docker kann man z.B. seine grafischen Anwendungen verwenden ohne Sie zu installieren, bzw ohne Sie in seinem Host-System zu installieren.
Um GUI-Anwendungen zu nutzen, muss Docker auf den X-Server des Linux oder Windows Host-Systems zugreifen können. Dazu gibt es Variablen und Ordner die dazu freigegeben werden müssen.
Docker Optionen Linux
Die DISPLAY Umgebungsvariable (bestimmt auf welchem X-Server eure GUI dargestellt wird)
-v DISPLAY=$DISPLAYFür die Kommunikation zwischen Anwendung und X-Server wird zusätzlich ein Unix-Domain Socket benötigt. Im Normalfall /tmp/.X11-unix/
-v /tmp/.X11-unix:/tmp/.X11-unixDamit der Software auch erlaubt wird auf eurem Display dargestellt zu werden, empfiehlt sich die Weiterleitung der .X-Authority Datei. Da die meisten Docker-Images mit einem root-Benutzer ausgestattet sind, ist das Ziel in diesem Beispiel /root/.Xauthority
-v $HOME/.Xauthority:/root/.Xauthority:rwManchmal reicht das nicht aus, und das Host-System muss mit einbezogen werden (funktioniert nur unter Linux). Mit der Angabe --network host wird dem Container mitgeteilt, seinen Netzwerklayer mit dem des Hosts zu ersetzen. Ports, die normalerweise im Container offen wären – sind damit im Host System geöffnet.
--network hostDocker Optionen Windows
Unter Windows besteht die Problematik zuerst einen X-Server zu bekommen. Ich selbst verwende dazu MobaXterm (extern: https://mobaxterm.mobatek.net/), das mir zusätzlich eine Shell bietet und man sich damit wie PuTTy auf remote-Host Maschinen anmelden kann. Alternativ dazu gibt es aber auch Xming.
Hat man MobaXterm gestartet, teilt einem der Xserver in der oberen rechten Ecke mit, auf welchem Display er gestartet wurde:

Das geben wir an das Docker-Image weiter
-e DISPLAY=192.168.x.x:0.0Beispiel: SplitsTree
Eine Quelle bioinformatischer Docker-Container bietet die Phenotypic Evolution Group (pegi3s) vom IBMC (Instituto de Biologia Molecular e Celular) aus Porto. (extern: https://pegi3s.github.io/dockerfiles/)
Als Beispiel verwende ich hier die Software SplitsTree als Docker Container:
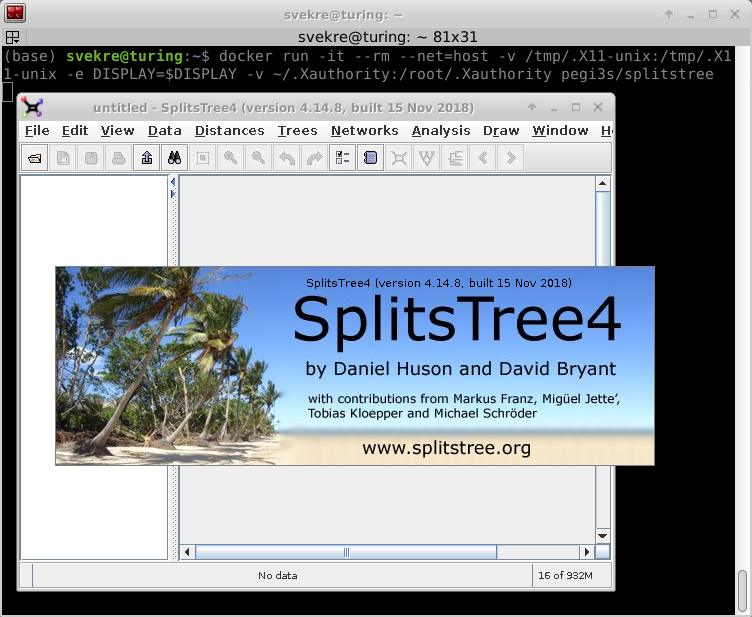
Splitstree im Selbstbau
Testweise habe ich mir ein Dockerfile gebaut, das bis zum herunterladen des Installations-Scripts von SplitsTree (Uni Tübingen) läuft, und danach Splitstree nur grafisch installiert werden muss.
# libglx 1.3 needed: therefore ubuntu.
FROM ubuntu:latest
MAINTAINER sven.kreienbrock@ruhr-uni-bochum.de
ENV DEBIAN_FRONTEND=noninteractive
RUN apt-get update && apt-get install -y wget git curl apt-transport-https \
vim default-jre libglx0
# SplitsTree Install
RUN mkdir /usr/src/SplitsTree
WORKDIR /usr/src/SplitsTree
RUN wget https://software-ab.informatik.uni-tuebingen.de/download/splitstree4/splitstree4_unix_4_16_1.sh
## how to run the install script?Dieses Dockerfile wurde dann wie folgt erstellt:
docker build -t svekre/splitstree .Nachdem ich dann SplitsTree installiert habe, wurde ein commit vom gegenwärtigen Containerstatus gemacht und das Image svekre/splitstree auf dockerhub.com gepusht.
Somit kann meine Version wie folgt geöffnet werden:
docker run -it --net=host -v /tmp/.X11-unix:/tmp/.X11-unix:rw -v $HOME/.Xauthority:/root/.Xauthority -e DISPLAY=$DISPLAY svekre/splitstree
Perl & autoadapt.pl
Ein anderes Problem ist z.B. das autoadapt.pl Skript für FastQC Sequenzdaten. Für das aktuelle Perl ist das Skript zu alt und mit FastQC gibt es noch eine Java-GUI Anwendung.
Hier braucht man zum einen eine alte Perl Version (mit multithreading) ohne seine eigene Perl Version zu beschädigen, sowie die Möglichkeit auf die GUI zurückzugreifen.
Vereint in einem Docker-Image kann das ganze ausgeführt werden:
docker run -it --net=host -v /tmp/.X11-unix:/tmp/.X11-unix:rw -v $HOME/.Xauthority:/root/.Xauthority -e DISPLAY=$DISPLAY svekre/perl:5.18.4
das gleiche, wenn wir es unter Windows starten:
docker run -it -e DISPLAY=192.168.x.x:0.0 svekre/perl:5.18.4